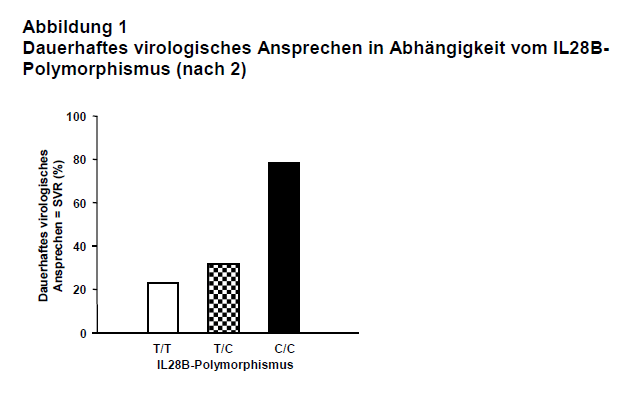
Die Standardtherapie der chronischen Infektion mit Hepatitis-C-Viren (HCV) besteht in der Kombination aus pegyliertem Interferon alfa (PEG-IFN alfa) plus Ribavirin (RBV). Sie führt zu dauerhaften virologischen Ansprechraten (SVR = Sustained Virological Response) von ca. 50% beim HCV Genotyp 1 und 80% beim Genotyp 2 und 3. In den aktuellen Leitlinien der Deutschen Gesellschaft für Verdauungs- und Stoffwechselkrankheiten wird eine Individualisierung der Therapie empfohlen, die von der Viruslast vor Therapiebeginn und vom Therapieansprechen innerhalb der ersten 4-12 Wochen abhängt (1). Die Therapie kann z.B. bei Patienten mit einer Genotyp-1-Infektion auf 24 Wochen verkürzt werden, wenn vor Therapie eine Viruslast < 600-800 Tsd. IU/ml besteht und wenn bereits nach vierwöchiger Therapie kein Virus mehr nachweisbar ist. Andererseits kann die Therapie bei Patienten, bei denen die Viruslast nach 12 Wochen nicht um > zwei Zehnerpotenzen abgefallen ist bzw. die noch eine Viruslast von > 30 Tsd. IU/ml haben, wegen mangelnder Erfolgsaussicht abgebrochen werden. Im Rahmen dieser Individualisierung der HCV-Therapie gewinnen neben dem Genotyp und der Höhe der Viruslast zusätzliche Kriterien an Bedeutung, wenn sie zusätzlich die Wahrscheinlichkeit eines Ansprechens auf die antivirale Therapie vorhersagen können. Kürzlich konnten in großen, unabhängigen Studien genetische Polymorphismen in der Nähe des IL28B-Gens, das für Interferon lambda (IFN lambda) kodiert, als wichtige und prädiktive Faktoren identifiziert werden. Z.B. fand sich bei Patienten mit chronischer Hepatitis C (Genotyp 1), die im Rahmen der IDEAL-Studie mindestens 12 Wochen lang mit der Kombination PEG-IFN plus Ribavirin behandelt wurden, dass die SVR mit einem bestimmten Polymorphismus assoziiert war (2). Eine SVR wurde etwa doppelt so häufig beobachtet (ca. 80%), wenn an einer bestimmten Region des IL28B-Gens Thymidin (T) durch Cytosin ersetzt war (C/C-Allel; s. Abb. 1). T/T- und T/C-Allelträger hatten hingegen nur SVR-Raten von ca. 40-50%. Der günstige C/C-Genotyp wird bei der weißen Bevölkerung deutlich häufiger als z.B. bei Afroamerikanern gefunden. Dieser Unterschied kann u.a. die deutlich schlechtere SVR bei Afroamerikanern erklären.
Die starke Assoziation zwischen Polymorphismen im Bereich des IL28B-Gens und hoher SVR konnte kürzlich in weiteren Studien bestätigt werden (2-5), u.a. auch bei Patienten mit einer Genotyp-2/3-Infektion (6). Hier war der IL28B-Polymorphismus vor allem bei den Patienten mit einer SVR assoziiert, die virologisch nicht rasch ansprachen (HCV-RNA < 50 IU/ml in der vierten Woche der Therapie). Die entsprechenden SVR lagen dann bei 87% (Genotyp C/C), 67% (T/C) und 29% (T/T) (6).
Zusätzlich konnte auch eine Assoziation mit einer Spontanheilung der Hepatitis C gezeigt werden, die beim günstigen C/C-Genotyp fast doppelt so häufig beobachtet wurde wie bei Patienten mit Genotyp T/T oder T/C (7). Eine noch stärkere Assoziation zeigte sich bei den Frauen der Anti-D-Kohorte aus der DDR (Infektion mit HCV Genotyp 1 im Rahmen einer kontaminierten Rhesusprophylaxe). Hier lag die Spontanheilung abhängig vom Genotyp bei 64% (C/C), 24% (T/C) oder nur bei 6% (T/T; 8).
Insgesamt deuten diese Ergebnisse auf eine wesentliche Bedeutung der Polymorphismen im Bereich des IL28B-Gens hin, sowohl im Ansprechen auf eine antivirale Therapie bei chronischer HCV-Infektion als auch im Spontanverlauf der akuten HCV-Infektion. Die zugrunde liegenden Mechanismen sind noch nicht bekannt. Es liegt aber nahe, dass eine Modulation der natürlichen Immunantwort gegen HCV beteiligt ist. So wurde z.B. eine antivirale Wirkung von IFN lambda in Zellkultur-Modellen gezeigt und in Phase-I-Studien bestätigt. Auch wenn der genaue Wirkmechanismus noch nicht geklärt ist, so steht doch mit der Bestimmung von Polymorphismen im Bereich des IL28B-Gens, die bereits von zahlreichen diagnostischen Laboren angeboten wird, ein wichtiger Prognosefaktor für das Ansprechen auf die Therapie bei chronischer HCV-Infektion zur Verfügung. In die aktuellen Leitlinien hat diese Untersuchung wegen der Aktualität noch keinen Eingang gefunden (1). Dennoch kann die Bestimmung eines IL28B-Polymorphismus bei Patienten mit chronischer HCV-Infektion, insbesondere bei HCV Genotyp 1, vor Beginn der Therapie erwogen werden. Denn einerseits ist der IL28B-Polymorphismus der stärkste Vor-Therapie-Prädiktor einer SVR bei Patienten mit einer Genotyp-1-Infektion (9). Andererseits stehen mit Telaprevir oder Boceprevir neue antivirale Substanzen für die Therapie der HCV-Infektion vor der Zulassung (2011/2012), die die Wahrscheinlichkeit einer SVR möglicherweise erhöhen (vgl. 10). Bei Patienten mit ungünstigem IL28B-Genotyp sollte daher die Indikation zur Therapie mit PEG-IFN alfa plus Ribavirin sehr sorgfältig überlegt werden und eventuell die Zulassung der neuen Substanzen abgewartet werden. Ob der IL28B-Polymorphismus auch zur Vorhersage des Ansprechens auf eine Tripeltherapie mit PEG-IFN alfa, RBV plus Telaprevir oder Boceprevir geeignet ist, muss in weiteren Studien geklärt werden. Eine erste kleine Studie aus Japan deutet aber auch hier auf eine wichtige prognostische Bedeutung des IL28B-Polymorphismus hin (11).
Fazit: Die Bestimmung des IL28B-Polymorphismus erlaubt eine bessere Vorhersage des spontanen Verlaufs und des Ansprechens auf eine antivirale Therapie mit pegyliertem Interferon alfa plus Ribavirin bei Hepatitis C. Ein dauerhaftes virologisches Ansprechen wird beim C/C-Genotyp etwa doppelt so häufig beobachtet (ca. 80%) wie beim Genotyp T/C oder T/T. Daher kann der IL28B-Polymorphismus neben Genotyp und Viruslast zur Abschätzung des Therapieerfolgs bei chronischer HCV-Infektion und damit zur genaueren des Patienten herangezogen werden.
Literatur
- Sarrazin, C., et al.: Z. Gastroenterol. 2010, 48, 289. Link zur Quelle Link zur Quelle
- Suppiah, V., et al. (IDEAL = Individualized Dosing Efficacy vs. flat dosing to Assess optimaL pegylated interferon therapy): Nat. Genet. 2009, 41, 1100. Link zur Quelle
- McCarthy, J., et al.: Gastroenterology 2010, 138, 2307. Link zur Quelle
- Rauch, A., et al.: Gastroenterology 2010, 138, 1338. Link zur Quelle
- Tanaka, Y., et al.: Nat. Genet. 2009, 41, 1105. Link zur Quelle
- Mangia, A., et al.: Gastroenterology 2010, 139, 821. Link zur Quelle
- Thomas, D.L., et al.: Nature 2009, 461, 798. Link zur Quelle
- Tillmann, H.L., et al.: Gastroenterology 2010, 139, 1586. Link zur Quelle
- Thompson, A.J., et al.: Gastroenterology 2010, 139, 1181. Link zur Quelle
- AMB 2010, 44, 68. Link zur Quelle
- Akuta, N., et al.: Hepatology 2010, 52, 421. Link zur Quelle